Es entsteht in der obersten Hautschicht – in den Zellen, die für die Färbung unserer Haut zuständig sind: das maligne Melanom, auch bekannt als schwarzer Hautkrebs. Gründe findet man in der genetischen Veranlagung oder äußeren Faktoren. Hauptrisikofaktor ist UV-Strahlung.
Sie kann Brüche in unserer DNA verursachen, gegen die unsere natürlichen Reparaturmechanismen manchmal nicht ankommen. Wenn Zellen entarten, das heißt sich unkontrolliert vermehren, und eine gewebeschädigende Masse bilden, spricht man dann von einem bösartigen Tumor. Man sollte also nicht nur an das namensgebende Tier denken, wenn man einen Sonnenbrand als krebsrot bezeichnet.
Warum genau Krebs entsteht, ist weitgehend noch ungeklärt. Antworten darauf liegen aber in unserer DNA.
Forschung an der THD
An der THD arbeitet die sechsköpfige Forschungsgruppe BIOinformatik unter der Leitung von Prof. Dr. Melanie Kappelmann-Fenzl. Sie ist zugleich Studiengangskoordinatorin des Bachelorstudiengangs Bioinformatik und des englischsprachigen Masterstudiengangs Life Science Informatics. Vor fünf Jahren hat sie begonnen Forschung und Lehre der Bioinformatik in Deggendorf zu etablieren. Ihr Gebiet verbindet die Erhebung und das Sammeln biologischer Daten mit computerbasierter Verarbeitung. So können Millionen von Datensätzen intelligent eingeordnet, strukturiert und verglichen werden.
Schwerpunkt der Forschungsgruppe ist das maligne Melanom – um genau zu sein, seine molekularbiologische Entstehung und Regulierung – vom Primärtumor, über das Eindringen in tiefere Gewebeschichten und sogar bis in die Blutbahn. Kappelmann-Fenzl hatte es sich dafür zum Ziel gemacht, Next Generation Sequencing (NGS) an die THD zu holen.
Crashkurs Genetik
Bei NGS handelt es sich um eine molekularbiologische Technologie, die es ermöglicht, die DNA auszulesen. Man spricht dabei von Gensequenzierung. Brauchte man 1990 buchstäblich 13 Jahre, um ein humanes Genom – die Gesamtheit der DNA eines Menschen – zum ersten Mal komplett auszulesen, schafft man es mit NGS praktisch über Nacht.
Man kann die DNA als eine Art Code betrachten, der alle genetischen Informationen zu unserem Körper, seinem Aufbau und seinen Funktionen beinhaltet. Relevant ist die Abfolge der Basenpaare, die wie metaphorische Sprossen einer Leiter die zwei Stränge der DNA verbinden. Ein menschliches Genom enthält etwa 3,2 Milliarden dieser Paare. Dabei finden sich aber nur die komplementären Basen Adenosin (A) und Thymin (T) sowie Cytosin (C) und Guanin (G) zusammen. Die Mechanismen, die diesen Code entschlüsseln und umsetzen, sind zugleich komplex und vielfältig.
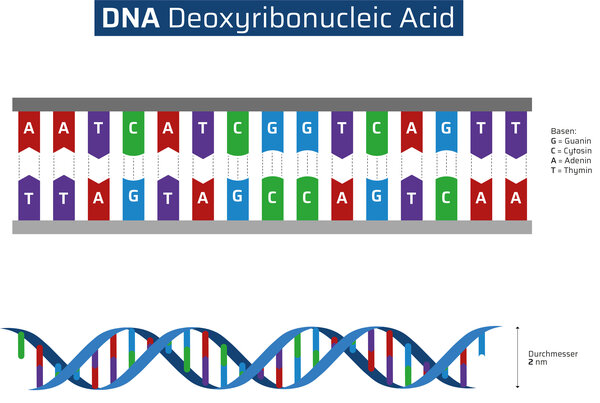
Aufbau der DNA: Basenpaare angeordnet auf einer Doppelhelix.
Im Fall des malignen Melanoms stehen besonders Zellwachstums- und Zellvermehrungsprozesse im Fokus. Eine zentrale Rolle kommt dabei der zweiphasigen Proteinbiosynthese zu. Sie ist der Prozess, der aus unserem „genetischen Bauplan“ Proteine erstellt, die die Funktion unserer Zellen regeln. Sehr vereinfacht heruntergebrochen verläuft sie so:
Phase 1 – Transkription: Damit die DNA den Zellkern nicht verlassen muss (wobei sie eventuell beschädigt werden könnte) wird sie kopiert. Ein Abschnitt mit spezifischem Informationsgehalt, besser bekannt als Gen, wird dazu zunächst entspiralisiert und in Einzelstränge aufgetrennt. An den Einzelsträngen ergibt sich durch die Anbindung von Basen eine komplementäre Kopie. Diese löst sich vom Original ab und transportiert die Informationen, wie ein Bote, aus dem Kern hinaus. Es handelt sich dabei um die mRNA, sie ist der Bauplan für Proteine.
Phase 2 – Translation: Aus dem Bauplan werden in der Zelle Ketten aus Aminosäuren hergestellt: die Proteine. Diese dienen dem Körper beispielsweise als Hilfsmittel, um Produkte zu erzeugen, die den Phänotyp, also das Aussehen, beeinflussen. Beispielsweise Farbstoffe für die Iris.
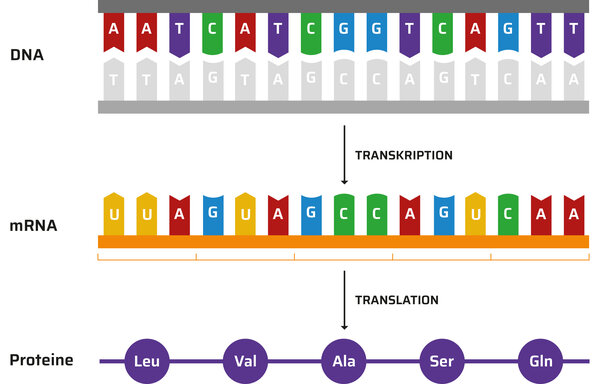
Proteinbiosynthese: Der Prozess, der aus unserem „genetischen Bauplan“ funktionsrelevante Proteine erstellt.
Entstehung maligner Melanome
Es gibt nicht die eine Zellmutation, die zu Krebs führt. Tumorzellen folgen ihren ganz eigenen Spielregeln. Und davon haben sie viele. Next Generation Sequencing liefert die Daten, um diese Vielfältigkeit zu ergründen.
Es konnten zwar bereits einige begünstigende Faktoren wissenschaftlich analysiert werden, aber noch nicht alle Veränderungen, die zur unkontrollierten Zellvermehrung von Tumoren führen. Der Bioinformatiker und wissenschaftliche Mitarbeiter Zubeir El Ahmad widmet sich in seiner Doktorarbeit zum malignen Melanom speziell Proteinen aus der AP1-Transkriptionsfamilie. AP ist kurz für Activating Protein. Diese wirken auf die Herstellung der mRNA, beinahe wie Schalter, die Gene aktivieren oder deaktivieren. El Ahmad analysiert dazu Daten, die mit NGS ausgelesen werden. Die Fragestellungen: Führt eine Fehlregulation durch AP1-Faktoren dazu, dass Gene stärker aktiviert werden, worauf unkontrolliertes Zellwachstum folgt? Und kann eine Störung dieser Proteine dazu führen, dass hunderte andere Gene fehlreguliert werden?
Was ist so „Next Generation“ an NGS?
Im Gegensatz zu älteren Generationen der Gensequenzierung, besticht es besonders durch zwei technologische Entwicklungen:
- Parallelsequenzierung: das Auslesen von Millionen von DNA-Fragmenten zur gleichen Zeit.
- Hohe Durchsatzkapazität: die Ausgabe großer Datenmengen in geringer Zeit.
Daraus ergibt sich neben der besseren Analysebasis auch ein finanzieller Vorteil. Wir erinnern uns: Ein komplettes Genom kann mittlerweile beinahe über Nacht ausgelesen werden – das erste Sequenzierungsprojekt brauchte hingegen 13 Jahre. Lagen die Kosten damals bei etwa 3,2 Millionen US-Dollar pro Genom, sind es heute um die 100 US-Dollar.
Aber wie funktioniert das genau? Verschiedene Hersteller bieten verschiedene technische Plattformen an. Ein weit verbreitetes Verfahren basiert darauf, einsträngige DNA-Fragmente auf einer Platte anzusiedeln und dort zu vermehren (=amplifizieren) bis Cluster aus vielen, gleichen Fragmenten entstehen. Danach werden die vier Basen hinzugegeben, die sich in den Clustern an die jeweiligen komplementären Basen binden sollen. Diese sind mit Fluoreszenzmarkern ausgestattet und geben ein zur jeweiligen Base passendes Signal ab. Das Lichtsignal wird vom Computer gelesen und als Code festgehalten. Die Zugabe der Basen passiert zyklisch, das bedeutet, dass der Computer das Lichtsignal nach jeder Anbindung erfasst, während sich Stück für Stück der komplementäre DNA-Strang aufbaut.

Ein weit verbreitetes NGS-Verfahren: Auf einer Platte werden einsträngige DNA-Fragmente zur Analyse angeordnet.
Wie geht es weiter?
Solange der Tumor noch nicht gestreut hat – also die entarteten Zellen noch keine Metastasen gebildet haben – kann eine operative Entfernung bereits ausreichen. Sollten Zellen aber bereits in die Blutbahn eingedrungen sein und sich im Körper verbreitet haben, kommt auch bei malignen Melanomen beispielsweise eine Chemotherapie zum Einsatz. Diese greift undifferenziert alle sich schnell teilenden Zellen im Körper an und dazu gehören neben Tumorzellen beispielsweise auch Haarzellen. Darum ist es essenziell, die molekularen Prozesse, die die onkogenen Wirkungen herbeiführen, punktgenau zu charakterisieren.
Dann kann zielgerichtet therapiert werden. Unter dem Begriff Targeted Therapy werden medikamentöse Methoden beschrieben, die nur an bestimmten biologischen Markern aktiv werden und den Rest des Organismus ungestört lassen. Für viele Krankheitsbilder ist das bereits Tagesgeschäft – Aspirin gegen Kopfschmerzen zum Beispiel – für Krebsarten besteht aber noch ein großes Potenzial.
Wo aber kommen die Gewebeproben dafür her? Verdächtige als auch unauffällige? Ohne die können schließlich keine Auswertungen, keine Statistiken vorgenommen werden. Dabei ist das Entfernen auffälligen Gewebes ein alltäglicher Diagnostikschritt, aber wer macht das ohne medizinische Begründung? Und wer darf Gewebeproben eigentlich zu Forschungszwecken nutzen?
An medizinischen Einrichtungen mit Lehr- und Forschungsauftrag, wie Unikliniken, ist der Zugang zu Biomaterial bedeutend einfacher als an einer technisch ausgerichteten Hochschule. Zudem erfolgen Einwilligungserklärungen von Patientinnen und Patienten meistens auf freiwilliger Basis. Die Forschungsgruppe an der THD hat deswegen vielzählige Kooperationen aufgebaut, beispielsweise mit der Molekularpathologie am Donauisar Klinikum in Deggendorf oder dem Institut für Biochemie an der Friedrich-Alexander-Universität in Nürnberg-Erlangen.
BIOinformatik an der THD
Die Forschungsgruppe BIOinformatik bringt Struktur in eine schier unendliche Flut aus Daten. Von der Sequenzierung über die Analyse bis zur Auswertung gilt es für die Forschenden, nicht nur komplexe biologische Zusammenhänge zu verstehen, sondern ebenfalls, diese mittels Informatik verständlich für Mediziner bereitzustellen. Nur so kann eine klare, aussagekräftige Datenlage zur Entwicklung von Behandlungsmethoden geschaffen werden.